Details
2024
Auch in diesem Jahr nahmen Schülerinnen und Schüler des Heisenberg-Gymnasiums am Regionalwettbewerb “Jugend forscht” in der DASA teil. Insgesamt stellten sieben Schülerinnen und Schüler vier Projekte aus dem Fachbereich Biologie vor.
Erstmals nahmen auch Schülerinnen der Klasse 10 an dem Wettbewerb teil. Sidra Al Maghrebi und Simona Hilla hatten für diesen Wettbewerb die Volumenproduktion von CO2 bei verschiedenen Hefen untersucht, Emilia Narozny und Amelie Schürmann aus dem Kombucha-Pilz eine vegane Lederalternative hergestellt. Emilia und Amelie gewannen den „Sonderpreis für nachwachsende Rohstoffe“.
Aus der Oberstufe gab es auch zwei Beiträge, Bastian Wiegel und Annina Böttger untersuchten einen Gordon Setter auf die Erbkrankheit „Progressive Retinaatrophie“, womit sie den „2. Platz im Bereich Biologie“ erhielten und Helena Bedenk entwickelte eine Teichmolch-Sonde für eine Multiplex realtime PCR. Helena gewann den „1. Platz im Bereich Biolgie“ und den „Sonderpreis der Deutschen Bundesstiftung Umwelt“. Der 1. Platz berechtigt sie zur Teilnahme am Landeswettbewerb Jugend forscht NRW vom 19. bis 21. März ´24 in Düsseldorf.
2023
Beim Regionalwettbewerb „Jugend forscht“ am 15.02.23 in der DASA konnte sich das Heisenberg-Gymnasium direkt 4-mal freuen.
Den 1. Platz und somit die Quailifikation für den Landeswettbewerb erhielten Darius Plumeyer und Leon Levers für ihre Methode zum Nachweis von Knoblauchkröten mit Hilfe von Gensonden.
Den 2. Platz erreichten Safae Kort und Baxos Ahmed mit ihrer Arbeit zum Nachweis von Insekten in Smoothies.
Den Sonderpreis Umwelt erhielten Armin Plumeyer und Tristan Böllecke.
2017
Analyse von eDNA zur Anwendung in der Ökologie
Kajeepan Kandepan, Jugend forscht Teilnahme
Die Idee: eDNA (enviromental DNA)
Wenn in der Forensik kleinste DNA Spuren einen Täter überführen, dann sollten doch auch alle Organismen DNA-Spuren in ihrem Lebensraum hinterlassen. DNA kann über diverse Wege in die Umwelt abgegeben werden. Zum Beispiel durch den Verlust von Zellen, Speichel oder Exkrementen von Lebewesen. In einigen Fällen kann auch die DNA von toten Lebewesen nachgew
iesen werden, jedoch hängt das mit der Zersetzungsrate zusammen, die wiederum von der Umwelt abhängig ist (HERDER et al. 2014, S. 15). Sollte eine Isolierung der eDNA gelingen, können Organismen in einem Lebensraum nachgewiesen werden ohne diese zu sehen. Gerade für seltene Tiere könnte dies den Schutz der Arten erleichtern.
2016
Nachweis einer Chitinase in Epipactis helleborine (L.) Crantz
und Ermittlung der zugehörigen Sequenz
Richard Pleuger, Jugend forscht Teilnahme 2016 (2. Platz Reginalwettbewerb)
Fragestellung:
Die Breitblättrige Stendelwurz (Epipactis helleborine (L) Crantz) gehört zur Gattung der Stendelwurzen (Epipactis) der Familie der Orchideen (Orchidaceae). Sie wird zwischen 20 und 100 cm groß und wächst krautig. Unter der Erde bildet sie ein Rhizom aus. Da sie wie die meisten Orchideen staubfeine Samen ausbildet, die frei von jeglichen, von der Mutterpflanze mitgegebenen, Nährstoffen ist, ist sie auf eine Versorgung durch Pilze angewiesen. Die Mykorrhiza geht bis zum reinen Parasitismus über, bei dem die Pflanze sich vom Pilz ernährt ohne ihm etwas im Gegenzug zu bieten.
Pilze bauen eine Art Zellwand auf. Allerdings ist diese aus Chitin, im Gegensatz zu pflanzlichen Zellen. Um Pilze effektiv verdauen zu können, müssen die Organismen Enzyme ausbilden können, welche das Chitin um die Pilzzellen verdaut. Solche Enzyme heißen Chitinasen. Chitinasen kommen bei allen Organismen auf, die in der Lage sind, Pilze zu verdauen.
Es stellte sich uns die Frage, ob man n in der Orchidee eine Chitinase nachweisen kann und sich die Sequenz bestimmen lässt, um so eine mögliches enzymatisches Fungizid herstelle zu können.
2015
Der Hybridkomplex Dactylorhiza majalis molekulargenetisch untersucht
Jannis Luncke, Jugend forscht Teilhame 2015 (2. Platz Landeswettbewerb)
Fragestellung
Neben der Polyploidisierung ist Hybridisierung ein h.ufiges Phänomen im Dactylorhizakomplex. Auf der untersuchten Orchideenwiese in Unna fanden wir einen solchen Hybriden. Nach morphologischen Merkmalen ließ sich dieser als Dactylorhiza x dinglensis (WILM.) SO. bestimmen. Demnach müsste es sich um einen Hybriden aus D. majalis und D. maculata handeln. Dabei stellte sich mir die Frage, ob sich diese Hybridisierung auch im genetischen Muster der ITSRegion wiederfinden l.sst. Dabei sollte zun.chst einmal geklärt werden ob sich die Methode des direct sequencing überhaupt zur Bestimmung von Elternarten von Hybriden eignet. Darüber hinaus entstand die Frage ob es sich bei D. dinglensis um eine tri- oder tetraploide Art handelt und damit ob Dactylorhiza maculata subs. maculata oder Dactylorhiza maculata subs. fuchsii einen Elternteil bilden. Zusätzlich wurde eine Fertilitätsprobe angefertigt, die die Frage nach der Anwendbarkeit des klassischen Artbegriffs zur Artbestimmung im Dactylorhiza komplex aufwarf.
Ergebnisse
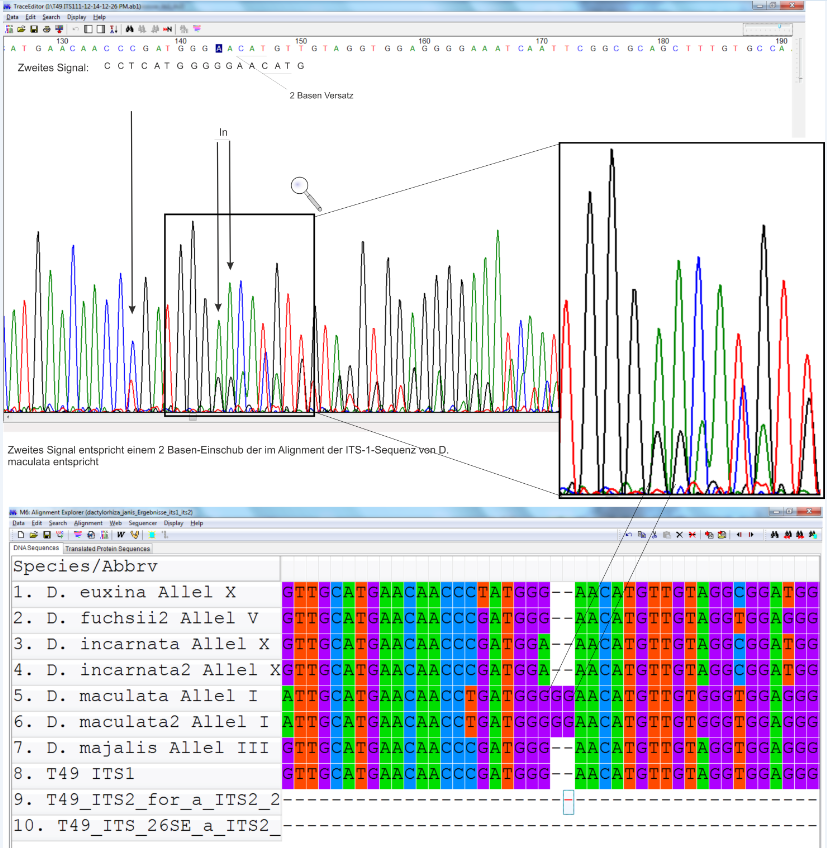
Offenbar handelt es sich bei Dactylorhiza dinglensis um einen Hybrid aus D. majalis und D. maculata. Für beide Gene lassen sich eindeutige Hinweise im Genom im Bereich der ITS-Region finden. Der Abgleich der Sequenzen mit der NCBI Datenbank ergab eine 100 Prozentige Übereinstimmung des Hauptsignals mit D. maculata. Auch sämtliche IN-DEL-Stellen stützen diese Annahme sowohl auf ITS1 als auch Auf ITS2 Jedoch stimmt die Sequenz auch weitestgehend mit D. fuchsii überein. Das Indel auf ITS2 zeigt jedoch das es sich um D. majalis handeln muss. Etwas aufwendiger war die Zuordnung des Untersignals. Das manuelle Ablesen aus dem Sequencer File ist verhältnismäßig fehleranfällig. Wichtig hierbei ist zu beachten, dass überall da kein Doppelsignal vorkommt, wo gleiche Basen an der selben Position stehen. Das liegt daran, dass der Laser nur Farben unterscheidet. Da gleiche Basen die selbe Farbe haben, entsteht nur ein Peak. Man muss also auch für das Untersignal dort Basen einfügen, wo nur ein Signal vorkommt. Auch findet sich ein durch IN-DEL-Stellen bedingter Versatz im Alignment. Trotzdem lassen sich die Basen gut ablesen. Auch das Ergebnis ist recht eindeutig. Die größte Übereinstimmung hat die Sequenz mit D. maculata. Sämtliche IN-DEL-Stellen bestätigen dies. Das Ergebnis bestätigt damit die Ergebnisse von PILLON et al. 2007. Bei dem als D. dinglensis bestimmten Hybriden handelt es sich tatsächlich um D. dinglensis.

2013
Trüffel-Suche mit Epipactis?
Sandra Scholz, Jugend forscht Teilnahme 2013 (Landeswettbewerb)
Eine Epipactis helleborine im Garten „macht“ noch keinen Trüffel, aber die Gattung Tuber kommt auch in NRW als Mycorrhiza-Partner bei Epipactis in Frage. Tuber rufum ist zwar kein Speise-Trüffel, aber es ist anzunehmen, dass auch andere Arten der Gattung Tuber eine Mycorrhiza mit Epipactis eingehen. Es wäre also von großem Interesse das Vorkommen der
Pilzpartner in einem größeren Gebiet zu untersuchen, um eine quantitative Verteilung zu bekommen. Problematisch ist allerdings, dass Epipactis helleborine eine geschützte Art ist, so dass diese nicht in der Natur gesammelt werden kann. Trotzdem könnte Epipactis helleborine eventuell als eine Art „Zeigerpflanze“ für mögliche Trüffelvorkommen genutzt werden, um dann gezielt direkt die Ectomycorrhizen der in Frage kommenden Bäume mit Hilfe der PCR zu bestimmen.
Des weiteren wäre es von großem Interesse die Frage des Epiparasitismus mit Hilfe von Epipactis zu untersuchen. Da einige Orchideen vollständig zur parasitären Lebensweise übergegangen sind und gar nicht mehr selber Fotosynthese betreiben, stellt sich die Frage, ob und wieweit Epipactis helleborine dazu in der Lage wäre. Vor allem da Epipactis helleborine zumindest in unserem Schulgarten bisher recht gut kultivierbar ist, können hier weitere Versuche zur Abhängigkeit entwickelt werden.
Zur Zeit versuchen wir einige Exemplare ohne Mycorrhizapartner wachsen zu lassen, indem diese in Granulattöpfe kultiviert werden um so vielleicht einen Partnerwechsel beschreiben zu können, bzw. eine pilzfreie Kultur. Auch hier könnte die Bestimmung des neuen Partner mit der hier beschriebenen Methode erfolgen.
2012
Vier auf einen Streich! Ist es möglich, Orchideen an Hand ihrer Allele mit Hilfe eines Arrays zu identifizieren?
Phillip Schulten, Besondere Lernleistung
Die DNA-Chip-Technologie ist ein seit dem Ende der 1980er Jahre entwickeltes Verfahren zur genetischen Untersuchung verschiedener DNA-Proben mit herausragender Nachweisempfindlichkeit. Wegen der hohen Anzahl an Tests, die in relativ kurzer Zeit mit vergleichsweise geringen Probenmengen möglich sind, hat sich dieses Verfahren etabliert. Vor der Entwicklung des Arrays mussten Proben mit Hilfe von elektrophoretischen Methoden oder chromatographischen Verfahren sehr zeitaufwändig untersucht werden. Diese Verfahren wiesen zudem eine hohe Fehlerquote auf.
Ich habe mir das Verfahren der DNA-Chips zur Nutze gemacht und einen Gentest entwickelt, der zuverlässig und innerhalb kurzer Zeit Aussage darüber trifft, ob bestimmte Dactylorhiza-Allele in verschiedenen Pflanzenproben vorhanden sind.
Einige Dactylorhiza-Arten unterscheiden sich nur geringfügig in ihrem genetischen Code und lassen phänotypisch nur eingeschränkt Unterscheidungen zu. Die Art Dactylorhiza majalis wird zwischen 30 und 70 cm groß und besitzt zumeist purpurrote Blüten. [Fletcher, 2004] Sie ist allotetraploid und wahrscheinlich aus den Arten Dactylorhiza fuchsii und Dactylorhiza incarnata hervorgegangen. [Bröker, 2011] Die Art Dactylorhiza maculata wird zwischen 20 und 60 cm groß und bildet weißlich rosa bis rosa Blüten aus, ihr Stängel ist markig. [Bröker, 2011]
Zuvor wurden die verschiedenen Arten sequenziert und manuell mit dem Computerprogramm „MEGA“ ausgewertet. So tat sich die Frage auf, ob die DNA-Chip-Technologie nicht auch auf dem Gebiet der Pflanzen Anwendung finden könnte, da dieses Verfahren bislang für Pflanzen weltweit noch nicht genutzt wurde. Die Entwicklung eines solchen Chips stellt somit ein gewisses Novum dar.












